Exercice 4 : Prise en main de BUGS & JAGS
Le projet BUGS (Bayesian inference Using Gibbs Sampling) a été initié en 1989 par l’unité de Biostatistique du MRC (Medical Research Council) de l’Université de Cambridge (au Royaume-Uni) afin de proposer un logiciel flexible pour l’analyse bayésienne de modèles statistiques complexes à l’aide d’algorithmes MCMC. Son implémentation la plus connue est WinBUGS, un logiciel clic-bouton disponible sous le système d’exploitation Windows. OpenBUGS est une implémentation fonctionnant sous Windows, Mac OS ou Linux. JAGS (Just another Gibbs Sampler) est une autre implémentation plus récente qui s’appuie également sur le langage BUGS. Enfin, il faut également noter le logiciel STAN, récemment développé à l’Université de Columbia qui n’est similaire à BUGS que dans son interface, s’appuyant sur des algorithmes MCMC innovants, comme par exemple les approches de Monte-Carlo Hamiltonien ou l’approche variationnelle. Une ressource très utile est le manuel de l’utilisateur de JAGS.
Afin de se familiariser avec le logiciel JAGS (et son interface depuis R via le package rjags), nous nous intéressons ici à l’estimation a posteriori de la moyenne et de la variance d’un échantillon de loi normale.
Charger le package
rjags.library(rjags)
Un modèle JAGS a 3 éléments :
- le modèle : spécifié dans un fichier externe (
.txt) selon la syntaxe propre àBUGS - les données : une liste contenant chaque données sous un nom correspondant à celui utilisé dans le modèle
- les valeurs initiales : (optionnel) une liste contenant les valeurs initiales pour les différents paramètres à estimer
Générer \(N=50\) observations selon une loi normale de moyenne \(m=2\) et d’écart-type \(s=3\).
N <- 50 # le nombre d'observations obs <- rnorm(n = N, mean = 2, sd = 3) # les observations (simulée)Enregistrer dans un fichier
.txtle modèle enBUGSdéfini à l’aide du code suivant :# Modèle model{ # Vraisemblance for (i in 1:N){ obs[i]~dnorm(mu,tau) } # A priori mu~dnorm(0,0.0001) # propre mais très plat (faiblement informatif) tau~dgamma(0.0001,0.0001) # propre mais très plat (faiblement informatif) # Variables d'interet sigma <- pow(tau, -0.5) }- Chaque fichier de spécification de modèle doit commencer par l’instruction
model{qui indique àJAGSqu’il s’apprête à recevoir la spécification d’un modèle. - Ensuite il faut mettre en place le modèle en bouclant (boucle for) sur l’ensemble des observations. Ici, nous voulons déclarer qu’il y a
Nobservations, et que pour chacune d’entre elleobs[i]suit une loi normale (caractérisée avec la commandednorm) de moyennemuet de précisiontau.
⚠️ Attention : dansBUGSla distribution Normale est paramétrée par sa précision, qui est l’inverse de sa variance (\(\tau = 1/\sigma^2\)). - Ensuite, il s’agit de définir les lois a priori pour nos deux paramètres
muettau, qui sont identiques pour chaque observation. Pourmu, nous prenons un a priori selon la loi normale de moyenne \(0\) et de précision \(10^{-4}\) (donc de variance \(10\,000\) : cela correspond à un a priori faiblement informatif et relativement étalé au vu de l’échelle de nos données). Pourtaunous prenons la loi a priori conjuguée dans un modèle Gaussien, c’est-à-dire la loi Gamma (avec des paramètres très faibles, là encore pour rester le moins informatif possible). - Enfin, nous donnons une définition déterministe de paramètre d’intérêt supplémentaire, ici l’écart-type
sigma.
NB:~indique une définition probabiliste d’une variable aléatoire, tandis que<-indique une définition par une formule déterministe.
- Chaque fichier de spécification de modèle doit commencer par l’instruction
À l’aide de la fonction
jags.model(), créer un objetjagsdansR.monpermierjags <- jags.model("normalBUGSmodel.txt", data = list("obs" = obs, "N" = length(obs)))## Compiling model graph ## Resolving undeclared variables ## Allocating nodes ## Graph information: ## Observed stochastic nodes: 50 ## Unobserved stochastic nodes: 2 ## Total graph size: 58 ## ## Initializing modelÀ l’aide de la fonction
coda.samples(), obtenir un échantillon de taille \(2\,000\) des distributions a posteriori des paramètres de moyenne et d’écart-type.res <- coda.samples(model = monpermierjags, variable.names = c("mu", "sigma"), n.iter = 2000)En étudiant le résultat obtenu, calculer les estimateurs de la moyenne a posteriori et de la médiane a posteriori pour
muetsigmaet donner un intervalles de crédibilité à 95% pour chacun.plot(res)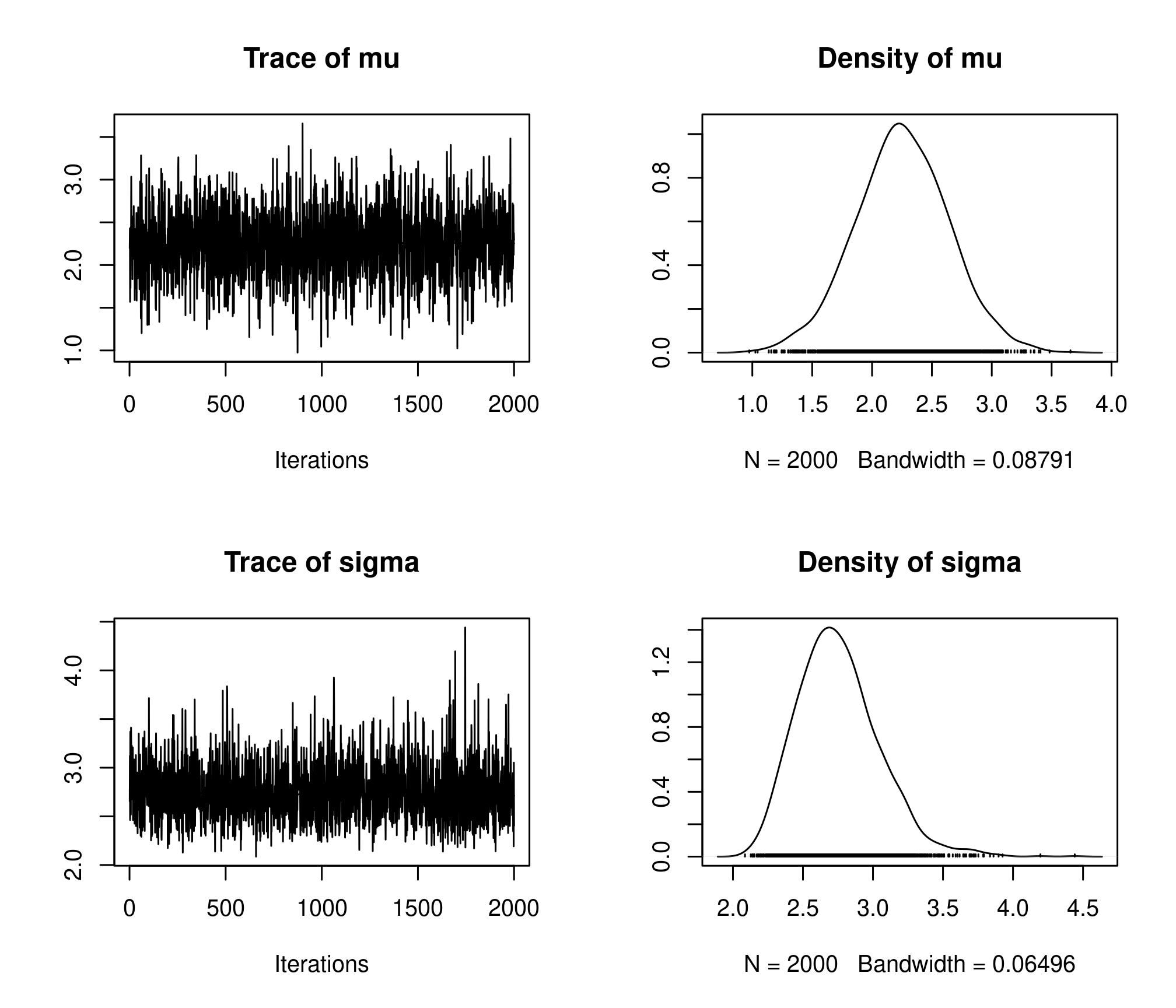
resum <- summary(res) resum## ## Iterations = 1:2000 ## Thinning interval = 1 ## Number of chains = 1 ## Sample size per chain = 2000 ## ## 1. Empirical mean and standard deviation for each variable, ## plus standard error of the mean: ## ## Mean SD Naive SE Time-series SE ## mu 2.251 0.3879 0.008675 0.008512 ## sigma 2.754 0.2923 0.006536 0.006536 ## ## 2. Quantiles for each variable: ## ## 2.5% 25% 50% 75% 97.5% ## mu 1.466 2.000 2.250 2.508 3.031 ## sigma 2.277 2.551 2.728 2.927 3.393resum$statistics["mu", "Mean"]## [1] 2.250648resum$statistics["sigma", "Mean"]## [1] 2.754404resum$quantiles["mu", "50%"]## [1] 2.249802resum$quantiles["sigma", "50%"]## [1] 2.728127resum$quantiles["mu", c(1, 5)]## 2.5% 97.5% ## 1.465933 3.031068resum$quantiles["sigma", c(1, 5)]## 2.5% 97.5% ## 2.276742 3.393362Charger le package
coda. Il s’agit d’un package contenant un certain nombre de fonctions pour le diagnostic de convergence des algorithmes MCMC et le traitement des résultats.library(coda)Afin de pouvoir diagnostiquer la convergence d’un algorithme MCMC, il est nécessaire de générer plusieurs chaînes de Markov différentes, partant de valeurs initiales différentes. Recréer un objet
jagsen spécifiant l’utilisation de 3 chaines de Markov parallèles et en initialisantmuettauà \((0; -10; 100)\) et \((1; 0,01; 0.1)\) respectivement (ProTip: utiliser unelistdelist— une pour chaque chaîne). Commenter.monjags2 <- jags.model("normalBUGSmodel.txt", data = list(obs = obs, N = N), n.chains = 3, inits = list(list(mu = 0, tau = 1), list(mu = -10, tau = 1/100), list(mu = 100, tau = 1/10)))## Compiling model graph ## Resolving undeclared variables ## Allocating nodes ## Graph information: ## Observed stochastic nodes: 50 ## Unobserved stochastic nodes: 2 ## Total graph size: 58 ## ## Initializing modelres2 <- coda.samples(model = monjags2, variable.names = c("mu", "sigma"), n.iter = 1000) plot(res2)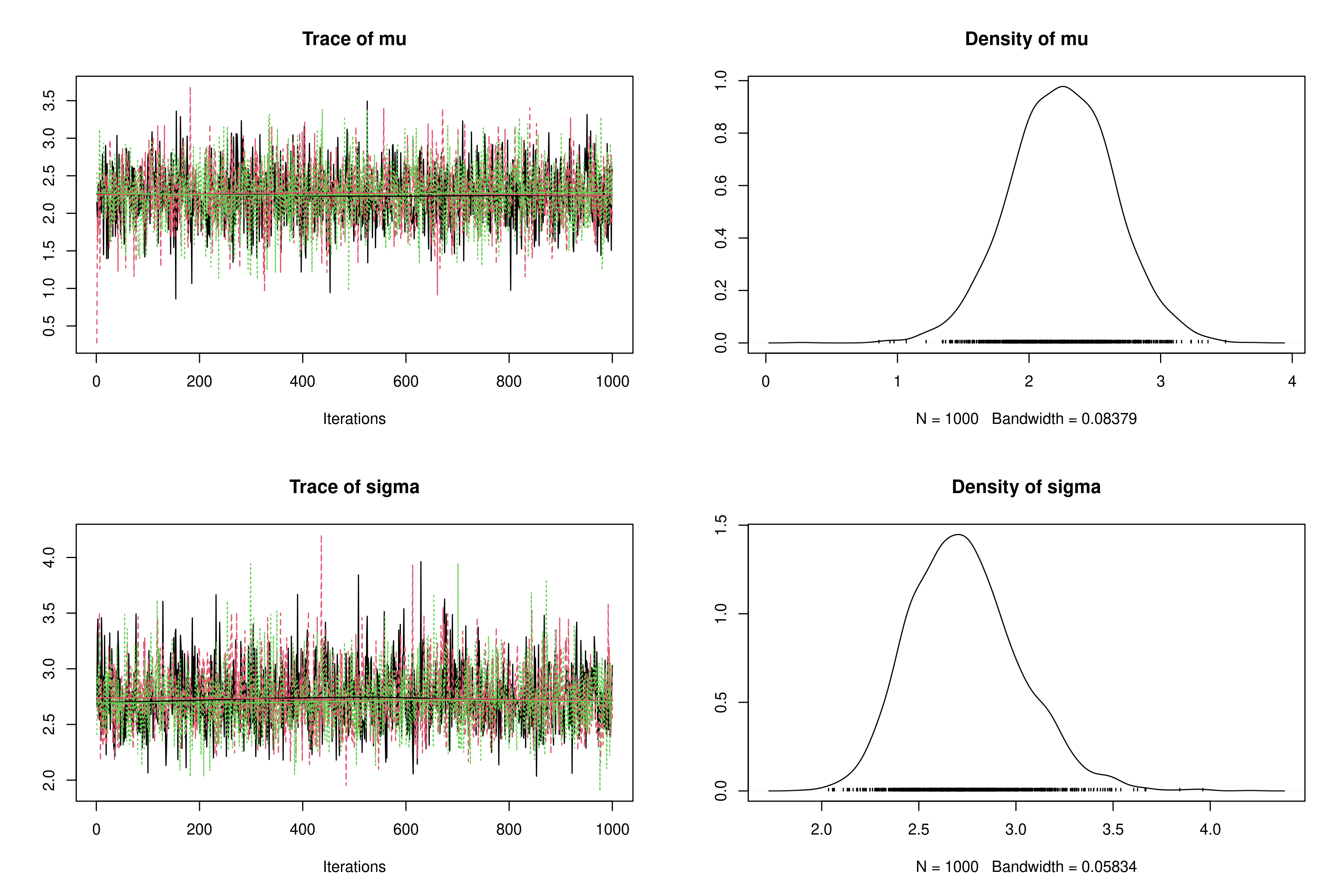
À l’aide de la fonction
gelman.plot(), tracer la statistique de Gelman-Rubin.gelman.plot(res2)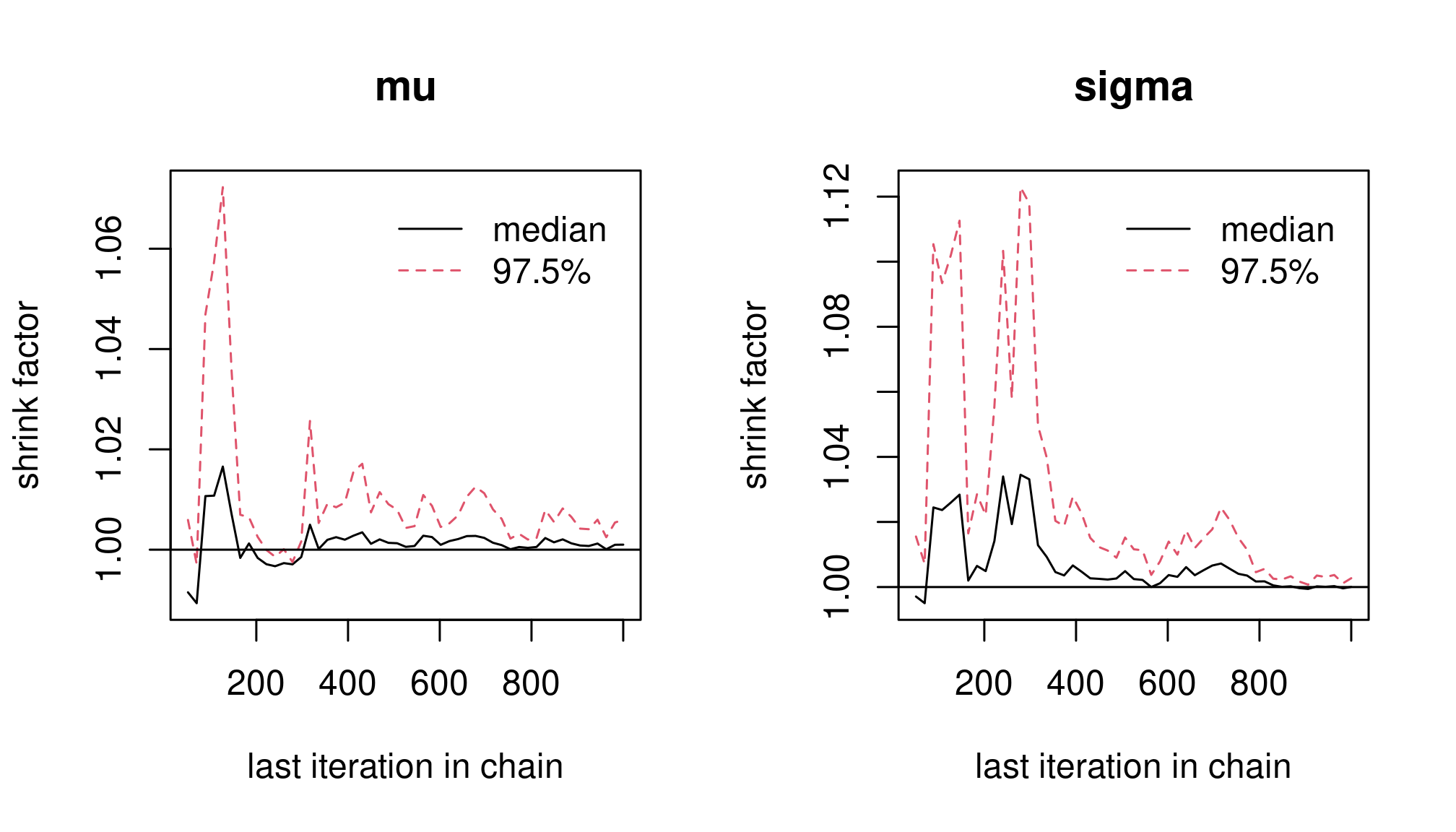
À l’aide des fonctions
autocorr.plot()etacfplot()évaluer l’auto-corrélation des paramètres étudiés.acfplot(res2)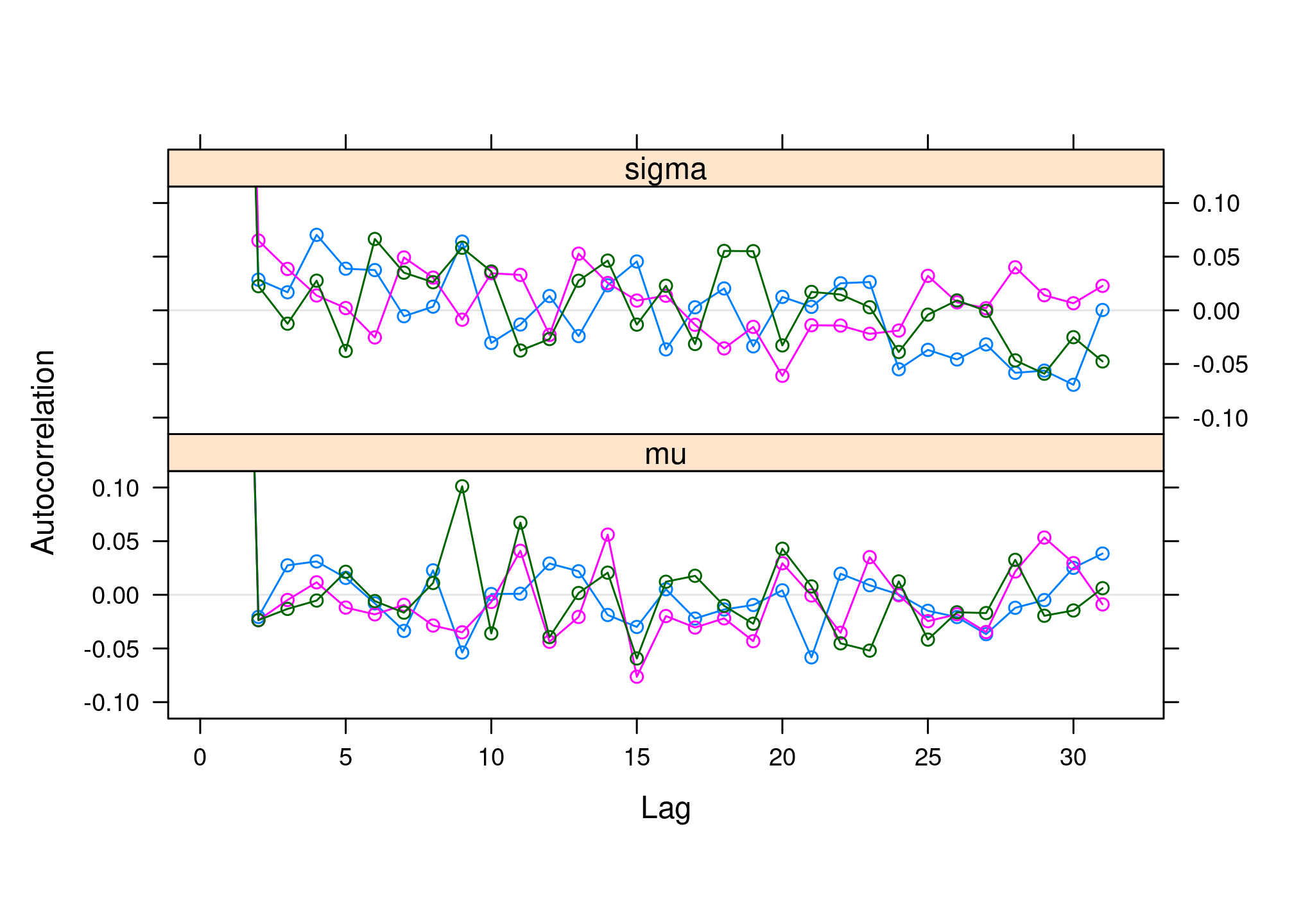
par(mfrow = c(3, 2)) autocorr.plot(res2, ask = FALSE, auto.layout = FALSE)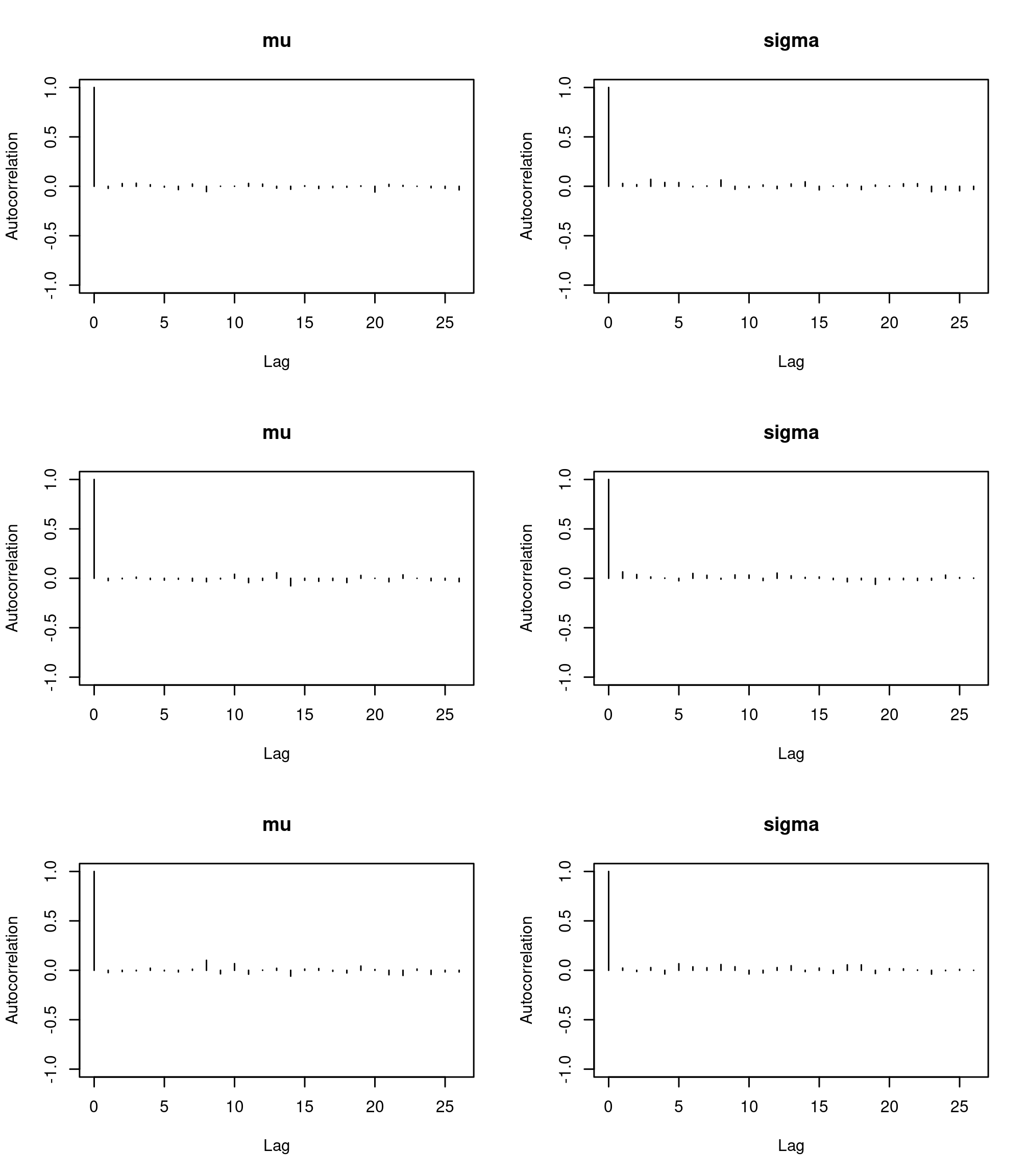
À l’aide de la fonction
cumuplot()évaluer les quantiles cumulés des paramètres étudiés. Comment les interpréter ?par(mfrow = c(3, 2)) cumuplot(res2, ask = FALSE, auto.layout = FALSE)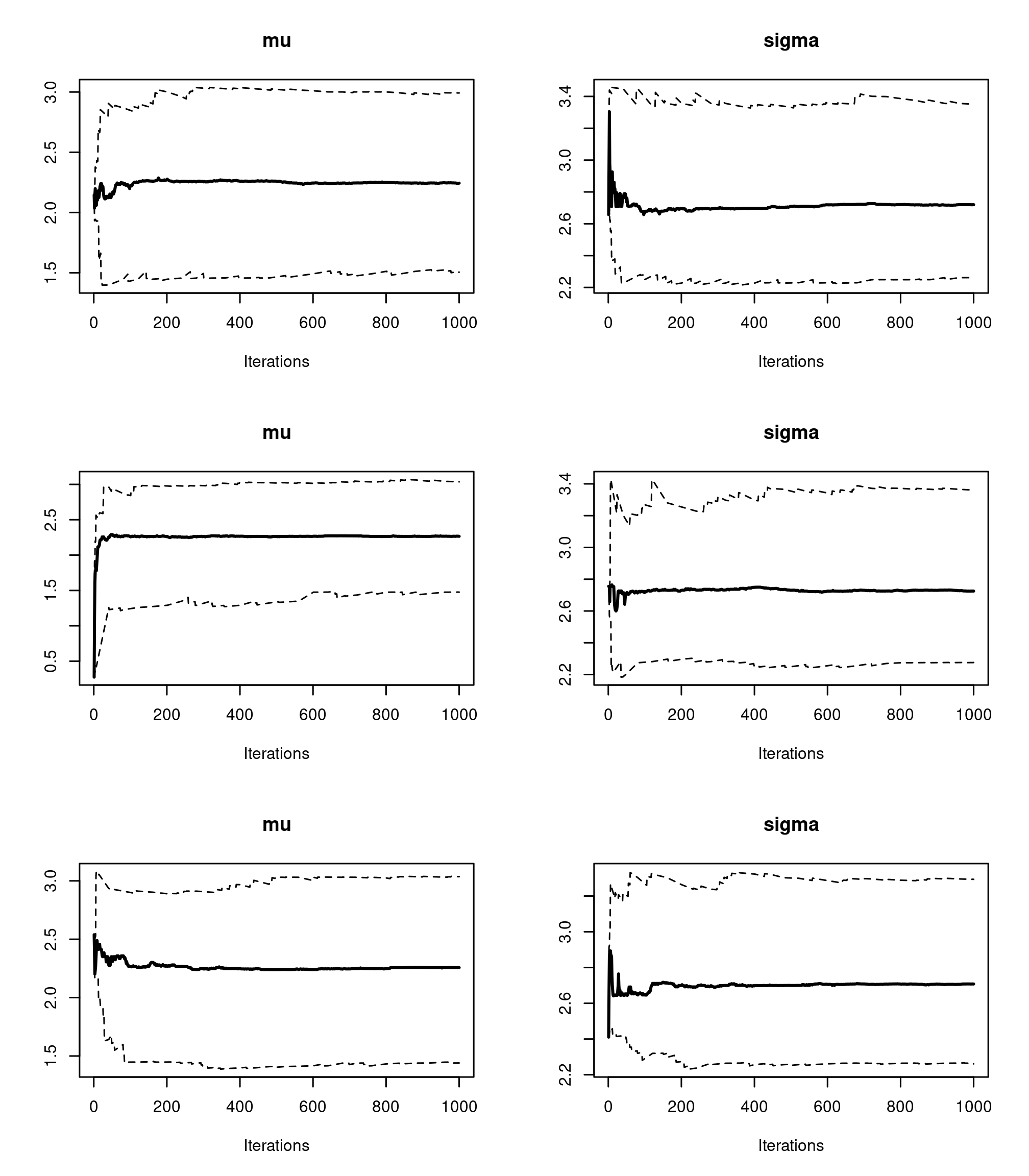
À l’aide de la fonction
crosscorr.plot()évaluer les corrélations entre les paramètres étudiés. Comment les interpréter ?crosscorr.plot(res2)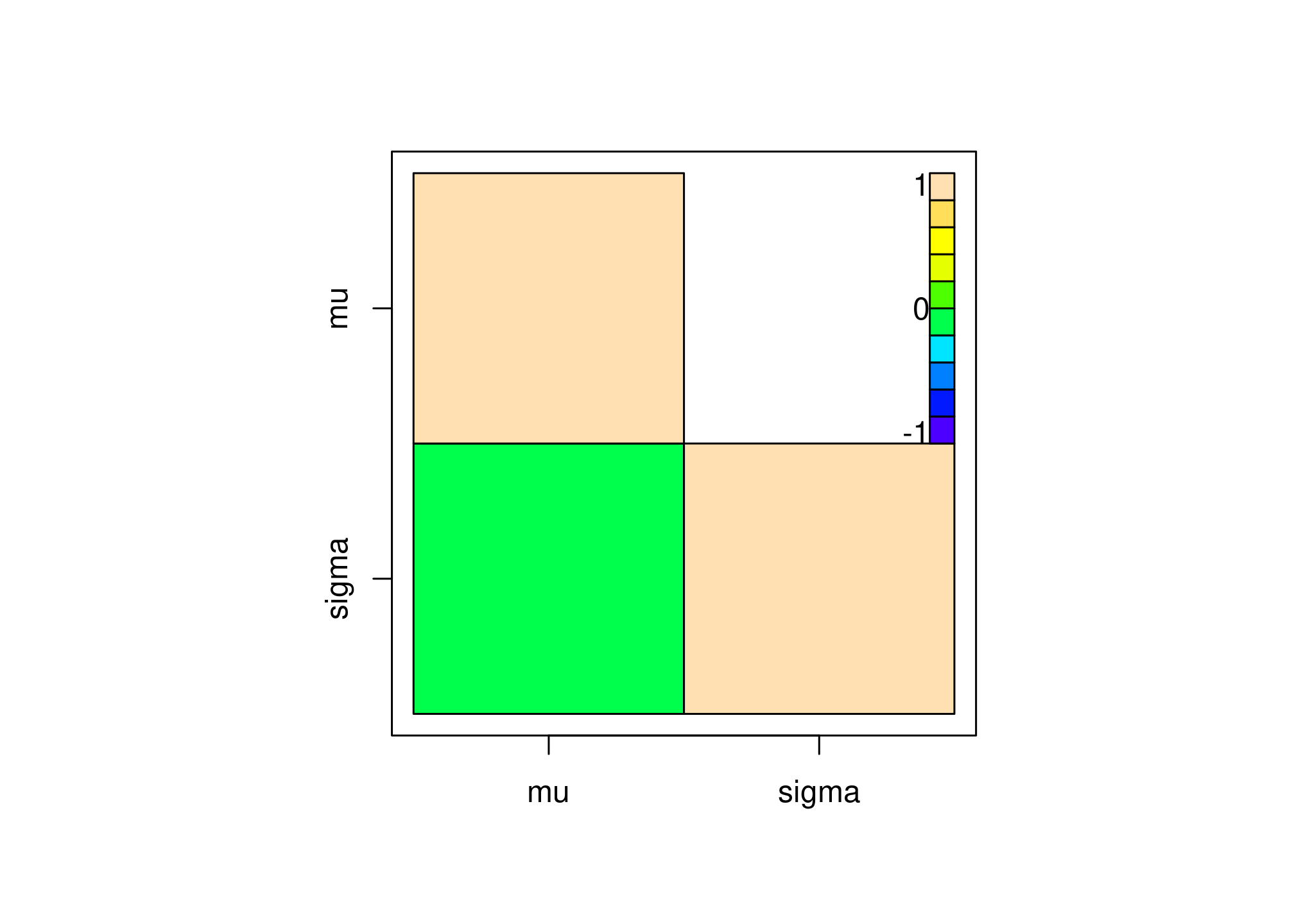
À l’aide de la fonction
hdi()du packageHDIntervaldonner les intervalles de crédibilités de plus haute densité a posteriori à 95%, et les comparer à ceux obtenus à partir des quantiles \(2,5\)% et \(97,5\)%.hdCI <- HDInterval::hdi(res2) hdCI## mu sigma ## lower 1.512342 2.248265 ## upper 3.054642 3.306796 ## attr(,"credMass") ## [1] 0.95symCI <- summary(res2)$quantiles[, c(1, 5)] symCI## 2.5% 97.5% ## mu 1.474634 3.025086 ## sigma 2.265202 3.335536symCI[, 2] - symCI[, 1]## mu sigma ## 1.550452 1.070334hdCI[2, ] - hdCI[1, ]## mu sigma ## 1.542300 1.058532